本文是一篇代写医学论文,本课题根据表达多倍化指数的结果,发现植物多倍化产生的亚基因组在表达水平上的差异总体较小,大部分的同源基因都不表达或表达无明显差异。但基因丢失的多倍化指数较小的植物其表达多倍化指数比其它植物更小,表明多倍化产生的亚基因组在基因表达上存在差异,表达多倍化指数在一定程度上能衡量亚基因组的表达差异。
第 1 章 绪论
1.1 植物多倍化的研究现状
与其它真核生物基因组不同,植物基因组的进化速率更快,不仅导致了植物基因组丰富的遗传多样性[1,2],同时也造成了植物基因组的不稳定性。这种不稳定性会导致植物基因组发生更频繁的变化,表现为基因重复(Gene duplication)和多倍化(Polyploidy)[3-9]。基因重复可以是单个基因或多个基因的重复,也可以是一条染色体片段或整条染色体的重复。多倍化是指全基因组水平的重复,也被称为全基因组加倍(Whole-genome duplications, WGDs)。发生过全基因组加倍的植物,理论上其染色体数目也会发生加倍形成多倍体,然而事实上,大部分的现存植物依然是二倍体,主要是因为植物全基因组加倍事件所产生的基因组往往不够稳定,在加倍事件后会发生大量的染色体重组(Chromosomes rearrangement)、DNA 颠倒(DNA segmental inversion)、DNA 片段的丢失(DNA segmental loss)等现象,从而使多倍体恢复到二倍体,这种现象被称为二倍化(Diploidization)[10,11]。植物多倍化往往不会发生一次,新一轮的多倍化过程可以在这样的二倍体中再次发生,并伴随着新一轮的二倍化,这种反复的循环伴随着植物的进化不断地出现,极大的增加了植物基因组的复杂性。古老的加倍事件导致植物基因组中产生大量的重复基因,而这些重复基因在植物抗病,抗逆境胁迫等方面都有一定的推动作用[12]。研究表明,植物在其两亿年的进化历史中多次发生了全基因组加倍事件,并且产生了大量的重复染染色体片段以及重复基因[13-17]。这种基因自身的不稳定性和多倍化之后产生的大量重复基因成为遗传创新的重要材料来源[18,19]。随着测序技术的发展,越来越多的物种基因组序列被鉴定,同时也提供了更加充分的证据证明了多倍化在陆生植物多样性[5,20-22]和作物驯化[23,24]方面的作用。
1.2 植物多倍化的研究方法
多倍化的研究依托于测序技术的飞速发展。随着高通量测序技术的出现,越来越多的植物全基因组序列信息被获取,而如何快速准确的从大量的基因组测序结果中分析其发生的多倍化事件,成为科学家们关注的问题。比较基因组学的出现为研究植物多倍化提供了大量的理论依据和技术手段。比较基因组学是一门生物学研究领域比较不同基因组特征,对基因组测序产生的基因进行比较,研究序列基本的生物学相似性和差异,对研究的物种之间的进化关系进行判断,了解基因功能、群体遗传和物种进化的学科。比较基因组学的基础是相关生物基因组的相似性。两种具有共同祖先的物种,他们基因组之间的差别是由祖先物种基因组进化而来,两个物种在进化阶段上越接近,它们的基因组相关性就越高。如果物种之间存在很近的亲缘关系,那么他们的基因组就会表现出共线性,即基因序列的部分或全部保守。基于比较基因组学,寻找基因组中的同源序列,区分序列之间的同源性关系,这样就可以根据已知的基因组信息去定位未知基因组中的基因,从而揭示基因潜在的功能,有助于我们理解多倍化过程与基因组结构以及基因功能进化的联系[49]。比较基因组学以计算机作为辅助手段,以解决生物学问题为目标,对多个物种间、物种内的基因组进行比较分析,解决传统生物学难以解决的问题,为生物进化机制的阐述提供了新的机会。
多倍化的研究以同源基因为基础,同源基因是指来源于共同祖先的相似序列。同源基因又可以分为直系同源基因(Ortholog),旁系同源基因(Paralog)和外旁系同源基因(Out-paralog)。直系同源基因发生在物种之间,是由于物种分化所引起的同源基因;旁系同源基因是指发生全基因加倍后,在一个物种内产生的同源基因,也称为种内旁系同源基因;外旁系同源基因是指由于全基因加倍所导致的物种之间的旁系同源基因,也称为种间旁系同源基因。通过对基因组数据的比较,可以获取到同源基因的相关信息,从而判断多倍化的发生与次数。
第 2 章 基因组共线性分析
2.1 数据材料
物种间的共线性分析,对于研究物种发生的多倍化事件有着重要的作用。本章通过比对物种间的同源基因,绘制同源基因点阵图,提取共线性基因片段,计算共线性基因片段的核苷酸同义替换率,最终构建物种间的共线性基因列表,为后续的分析提供了基础。
图 2 展示了本课题所研究植物之间的系统发育关系及该物种所发的多倍化事件。
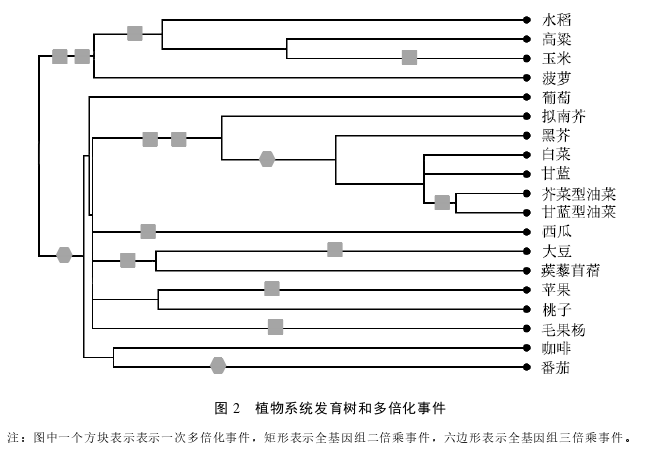
在物种进化研究中,通常会选取一个或者几个物种作为外类群来对所要研究的物种进行比较分析。基因组结构比较完整,与研究物种亲缘关系比较近,与研究物种分化后没有再单独发生过加倍事件的植物可作为研究物种的外类群。杨柳科、十字花科、蔷薇科、葫芦科、豆科和茄科植物在经历了双子叶全基因组加倍事件后又单独发生过加倍事件。本课题选取每个科的代表性物种进行研究,包括毛果杨(杨柳科),苹果(蔷薇科),西瓜(葫芦科),蒺藜苜蓿(豆科),番茄(茄科)。其中,毛果杨,西瓜,蒺藜苜蓿的外类群为葡萄,苹果的外类群为桃,番茄的外类群为中粒咖啡。其中,大豆在与蒺藜苜蓿分化后又单独经历了多倍化事件,以蒺藜苜蓿作为外类群,对大豆进行研究。芸薹属在与拟南芥分化后,又单独经历了多倍化事件,以拟南芥作为外类群,选取白菜,甘蓝,黑芥作为研究对象进行研究。白菜和甘蓝杂交,形成甘蓝型油菜,以白菜和甘蓝作为外类群,对甘蓝型油菜进行研究。白菜和黑芥杂交,形成芥菜型油菜,以白菜和黑芥作为外类群,对芥菜型油菜进行研究。在单子叶植物中,本课题选取了代表性的禾本科植物作为研究对象,禾本科植物与菠萝分化后,单独发生过一次多倍化事件,以菠萝作为外类群,对水稻进行研究。玉米在经历了禾本科植物共有的多倍化事件后,单独发生过多倍化,以高粱作为外类群,对玉米进行研究。
2.2 研究方法
2.2.1 数据下载与处理
本课题研究涉及的物种数据均来源于公共数据库及已发表文献,具体的基因组信息如表 1 所示。从数据库下载所需的基因组数据主要有,全基因组序列,基因组注释文件(General feature format, GFF), 基因编码序列(Coding sequence, CDS)和基因翻译蛋白文件(Peptide, PEP)。对于下载的数据利用 Perl、Python 等计算机语言处理成所需要的格式,便于后续研究分析使用。真核生物基因组中存在大量的可变剪切,导致一个基因会生成多个 mRNA 序列,因此,首先对 CDS 序列长度进行统计,得到每个基因对应的最长CDS序列的ID。然后根据ID对基因组注释文件(GFF)进行筛选,过滤没有定位到染色体上的基因相关信息,保留所有与 ID 对应的基因信息。将过滤后基因组注释文件处理成格式为包含染色体号、基因起始终止位置、基因转录方向、重新命名的基因 ID、原 ID、以及基因对应在染色体上的位置顺序的新文件。根据新文件对 CDS 和 PEP 进行过滤并修改 ID,便于后续的分析。
2.2.2 基因组间同源比对
将所选物种经过处理的 PEP 蛋白质文件与所选定外类群物种的 PEP 蛋白文件进行同源基因比对,使用 BLASTP[50]工具进行同源性比对打分,通过设置限制同源性条件,期望(E-value)限定为 1e-5,来获得输出格式为 TABULAR 的比对结果。TABULAR 格式的结果从左至右依次表示查询序列 ID 标识(Query id),比对上的目标序列 ID(Subject id),序列比对的一致性百分比(Identity),符合比对的比对区域长度(Alignment length),比对区域的错配数(Mismatches),比对区域的空位数目(Gap openings),比对区域在查询序列上的起始位点(Q. start),比对区域在查询序列上的终止位点(Q. end),比对区域在目标序列上的起始位点(S. start),比对区域在目标序列上的终止位点(S. end),比对结果的期望值(E-value),比对结果的打分值(Bit score)。
第 3 章 基因丢失的多倍化指数应用 .................................. 20
3.1 数据材料 ............................... 20
3.2 研究方法 ................................... 20
第 4 章 表达多倍化指数的开发与应用 ............................... 25
4.1 数据材料 ........................................ 25
4.2 研究方法 .............................. 25
第 5 章 甲基化多倍化指数的开发与应用 .................... 33
5.1 数据材料 ........................... 33
5.2 研究方法 ............................. 33
第 5 章 甲基化多倍化指数的开发与应用
5.1 数据材料
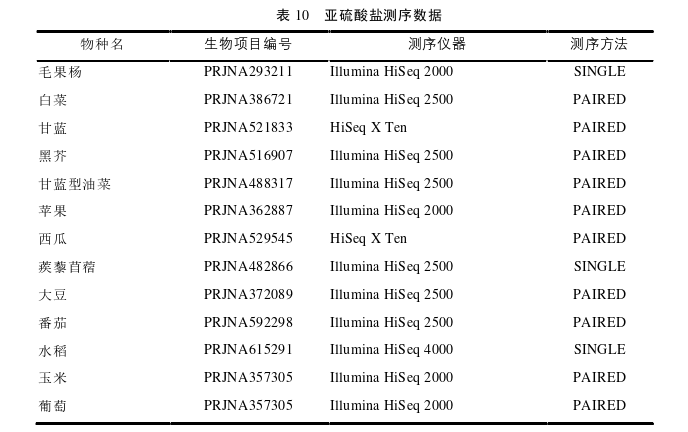
全基因组甲基化测序(Whole genome bisulfite sequencing, WGBS)的研究对象为基因组上的甲基化位点,本课题使用的亚硫酸盐测序(Bisulfite sequencing, BS-seq)数据均来自 NCBI(National center for biotechnology information)的 SRA(Sequence read archive)数据库,详细来源见表 10。
结论
本课题选取玉米等 14 个代表性的植物,开发了基因表达多倍化指数与甲基化多倍化指数两个统计量,并结合基因丢失的多倍化指数,从基因保留丢失、基因表达和甲基化 3 个方面,对植物多倍化进行分析,阐明了多倍化产生的亚基因组在不同方面的统计学差异。
根据表达多倍化指数的结果,发现植物多倍化产生的亚基因组在表达水平上的差异总体较小,大部分的同源基因都不表达或表达无明显差异。但基因丢失的多倍化指数较小的植物其表达多倍化指数比其它植物更小,表明多倍化产生的亚基因组在基因表达上存在差异,表达多倍化指数在一定程度上能衡量亚基因组的表达差异。
根据甲基化多倍化指数的结果,发现植物多倍化产生的亚基因组在甲基化水平上的差异总体也比较小,但与表达数据不同,大部分的同源基因其甲基化率都在其中一个基因中更高,但其在亚基因组间的分布却没有明显的偏好性。基因丢失的多倍化指数和表达多倍化指数小的植物其甲基化多倍化指数也相对更小,表明多倍化产生的亚基因组在基因甲基化率上也存在差异,甲基化多倍化指数在一定程度上能衡量亚基因组的甲基化差异。
利用这两个统计量来检验几千万年前四倍体化产生的玉米亚基因组之间的表达和甲基化差异。我们发现玉米亚基因组之间在这两个生物学特征上有轻微的差异。这些发现表明,玉米亚基因组有很大的不同,不仅是在基因丢失的层面上,而且在表达和甲基化的层面上也有不同。一致的是,一组玉米亚基因组优先保留了更多的基因,在表达水平上轻度占优,并受到更多 DNA 甲基化的影响。
参考文献(略)
